-
<통계학> t검정(t-test) - 2 (with Python)데이터 분석 관련 공부 2024. 1. 24. 21:40
https://jinhyunbae.tistory.com/129
<통계학> t검정(t-test) - 1
t검정이란 검정통계량이 귀무가설 하에서 t-분포를 따르는 통계적 가설검정이다. 어느 특정한 집단의 평균의 값을 추정하거나 두 집단의 평균 차이를 검정할 때 사용할 수 있다. t분포란? t분포
jinhyunbae.tistory.com
위 링크에서 설명한 t검정을 python의 scipy라이브러리로 구현해보자
데이터는 R에서 제공하는 cat 데이터를 csv 파일로 저장한 것이다.
https://github.com/vincentarelbundock/Rdatasets/blob/master/csv/MASS/cats.csv
데이터 불러오기
우선 사용할 라이브러리를 import하고 데이터를 불러온다.
import numpy as np import pandas as pd import scipy.stats as stats import matplotlib.pyplot as plt cats = pd.read_csv('./data/cats.csv') display(cats)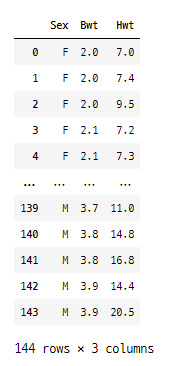
데이터는 144행 3열(성별, 몸무게, 크기)로 이루어져 있고, 성별은 범주형(M, F)변수이며
몸무게와 크기는 연속형 변수이다.
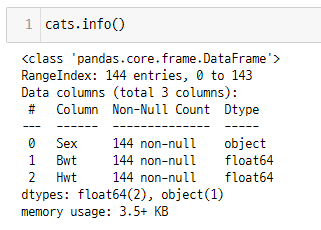
Null 값은 따로 존재하지 않기 때문에 그대로 데이터를 사용하였다.
일표본 t검정(One sample t-test)
일표본 t검정의 가설
- H0 : 고양이의 평균 몸무게는 2.6이다.
- H1 : 고양이의 평균 몸무게는 2.6이 아니다.
정규성 검정(Normality test)
우선 일표본 t 검정을 하기 위해서는 정규성 가정을 만족해야한다.
가정을 만족하는지 알아보기 위해서 정규성 검정을 실시하는데
정규성 검정에는 여러 가지가 있지만 많이 알려진 2가지를 적용해보았다.
첫 번째는 Shapiro-Wilk 검정이다. Shapiro-Wilk는 표본 수가 적을 때도 적용이 가능하도록 검정된 기법이다.
from scipy.stats import shapiro shapiro(cats['Bwt']) # 정규성 검정 결과 정규분포한다는 귀무가설을 기각ShapiroResult(statistic=0.9518786668777466, pvalue=6.730248423991725e-05)두 번째는 Kolmogorov-Smirnov 검정이다. Kolmogorov-Smirnov는 표본 수가 많을 때(수 천개) 사용하는 검정기법이다.
from scipy.stats import kstest kstest(cats['Bwt'], 'norm') # 정규성 검정 결과 정규분포한다는 귀무가설을 기각KstestResult(statistic=0.9772498680518208, pvalue=5.089961007561737e-237)두 정규성 검정 실행 결과를 보면 p-value가 유의수준 0.05보다 훨씬 낮아 표본의 분포는 정규성을 만족한다는 귀무가설을 기각하는 것을 볼 수 있다.
따라서 모수검정을 할 수 없기 때문에 비모수 검정 방법인 Wilcoxon 부호순위 검정을 이용해야한다.
mu = 2.6 # 검정하고 싶은 고양이 무게 평균 2.6kg # stats.wilcoxon(data.variable - data.mean(), alternative='greater or less or two_sided') stats.wilcoxon(cats.Bwt - mu, alternative='two-sided')WilcoxonResult(statistic=3573.0, pvalue=0.02524520294814093)검정 결과 p-value가 0.025로 유의수준 0.05보다 낮기 때문에 귀무가설을 기각한다. 따라서 고양이의 몸무게는 2.6이 아니라는 대립가설을 채택한다.
정규성을 만족한다는 가정하에 일표본 t-test도 돌려보았다.
stats.ttest_1samp(cats.Bwt, popmean=mu)Ttest_1sampResult(statistic=3.0564867998078107, pvalue=0.0026730362561723617)검정 결과 p-value가 0.002로 유의수준 0.05보다 낮기 때문에 귀무가설을 기각한다. 따라서 고양이의 몸무게는 2.6이 아니라는 대립가설을 채택한다.
독립표본 t검정(Independent sample t-test)
독립표본 t검정의 가설
- H0 : 고양이의 성별에 따른 몸무게 차이가 없을 것이다.
- H1 : 고양이의 성별에 따른 몸무게 차이가 있을 것이다.
female_cats = cats.loc[cats.Sex=='F', 'Bwt'] male_cats = cats.loc[cats.Sex=='M', 'Bwt']독립표본 t검정은 두 독립된 집단의 평균 차이를 비교하는 검정이므로 데이터를 암컷 고양이와 수컷 고양이의 몸무게로
분할하였다.
우선 정규성을 만족한다고 가정한 상태에서 모수검정을 먼저 시행해보겠다.
그리고 등분산 가정을 만족하는지를 알아보기 위해 levene의 등분산 검정을 실시하였다.
# 등분산 검정 stats.levene(female_cats, male_cats)LeveneResult(statistic=19.43101190877999, pvalue=2.0435285255189404e-05)등분산 검정 결과 p-value가 유의수준 0.05보다 낮게 나타나 등분산 가정이 기각되었음을 알 수 있다.
따라서 equal_var 파라미터를 False로 지정해주고 독립표본 t검정을 시행하였다.
이렇게 되면 Student t검정이 아니라 Welch t검정을 수행하게 되는 것이다.
stats.ttest_ind(female_cats, male_cats, equal_var=False, alternative='two-sided')Ttest_indResult(statistic=-8.70948849909559, pvalue=8.831034455859356e-15)검정 결과 p-value가 유의수준 0.05보다 낮기 때문에 귀무가설을 기각한다. 따라서 고양이의 성별에 따른
몸무게 차이가 있다고 말할 수 있다.
이제는 비모수 검정을 알아보자
from scipy.stats import shapiro print(shapiro(female)) print(shapiro(male)) from scipy.stats import ks_2samp print(ks_2samp(male, female))ShapiroResult(statistic=0.8909613490104675, pvalue=0.0003754299250431359) ShapiroResult(statistic=0.9788321852684021, pvalue=0.11895745247602463) KstestResult(statistic=0.548146523360386, pvalue=2.8636986115770924e-09)검정 결과 female의 몸무게가 정규성을 만족하지 않는 것으로 나타났다. 따라서 비모수 검정을 해주어야하는데
Man Whitney U검정이라고도 불리는 Wilcoxon 순위 합 검정을 시행하면 된다.
from scipy.stats import mannwhitneyu stats.mannwhitneyu(female, male)MannwhitneyuResult(statistic=757.5, pvalue=8.200502234321752e-11)검정 결과 마찬가지로 p-value가 0.05보다 낮기 때문에 귀무가설을 기각한다.
대응표본 t검정(One sample t-test)
data = {'before':[7,3,4,5,2,1,6,6,5,4], 'after':[8,4,5,6,2,3,6,8,6,5]} data = pd.DataFrame(data) display(data)대응표본에 관련된 데이터는 따로 없어서 그냥 생성해주었다.
대응표본은 두 집단이 동일한 집단이므로 등분산 가정은 만족한다고 보고 정규성 가정을 만족시키면 되는데
정규성 가정을 만족한다고 보고 진행하겠다.
대응표본 t검정의 가설
- H0 : 처치 전 후 집단 평균에는 차이가 없을 것이다.
- H1 : 처지 전보다 후의 집단 평균이 클 것이다.
위의 독립 표본 t검정의 가설과 차이가 있다면 대립 가설이 클 것이다로 한 방향을 지정하고 있다는 것이다.
이 경우에는 단측검정을 실시하는데 ttest_rel함수의 alternative 파라미터를
'two-sided' 가 아닌 'greater'로 지정해주면된다.
stats.ttest_rel(data['before'], data['after'], alternative='greater')Ttest_relResult(statistic=-4.743416490252569, pvalue=0.9994730643714917)검정 결과 p-value가 0.5보다 작기 때문에 귀무가설을 기각한다. 따라서 처치 전후 평균 차이는 통계적으로 유의하고
처치 후의 값이 더 커졌다고 할 수 있다.
정규성을 만족하지 않는 경우에는 Wilcoxon 부호 순위 검정을 해주면 된다.
stats.wilcoxon(data['before'], data['after'], zero_method='wilcox', alternative='greater')WilcoxonResult(statistic=0.0, pvalue=0.9958503920023596)'데이터 분석 관련 공부' 카테고리의 다른 글
<통계학> 분산분석(ANOVA) - 2 (One way ANOVA with Python) (1) 2024.01.25 <통계학> 분산분석(ANOVA) -1 (One-way-ANOVA) (2) 2024.01.25 <통계학> t검정(t-test) - 1 (1) 2024.01.23 <통계학> 통계적 가설 검정 -2 (신뢰수준, 1종 오류, 2종 오류) (0) 2024.01.23 <통계학> 통계적 가설 검정 -1 (귀무가설, 대립가설, p-value) (1) 2024.01.22